Задача
«Великая книга природы написана математическими символами».
Галилео Галилей
Красные стрелки соответствуют ингибированию, зеленые – активации. Вершины графа в виде многоугольника содержат несколько генных продуктов, овалы – только один ген.
Для молекулярного пути на можно определить коэффициенты влияния каждого гена на биологический эффект пути, учитывая тип взаимодействия генных продуктов и их комплексов друг с другом и выполняя обход графа по следующим правилам. Стартовая вершина – та, из которой можно прийти в наибольшее количество вершин (граф направленный). Вариантов стартовых вершин может быть несколько. Нужно выбрать из них любую, при которой сохраняется биологический эффект пути.
Стартовая вершина в начале обхода имеет коэффициент единицу, остальные равны нулю. При назначении коэффициентов граф считать ненаправленным. При прохождении через стрелку активации следующая вершина наследует коэффициент от предыдущей, а через стрелку ингибирования – наследует с обратным знаком.
Транскриптомную активацию пути можно измерить количественно в виде суммы двоичных логарифмов отношений уровня экспрессии в опухоли к уровню в норме. При этом, для каждого отношения должен быть учтен коэффициент влияния гена на эффект пути (коэффициент для гена наследуется от вершины).
При секвенировании РНК получены следующие уровни генной экспрессии:
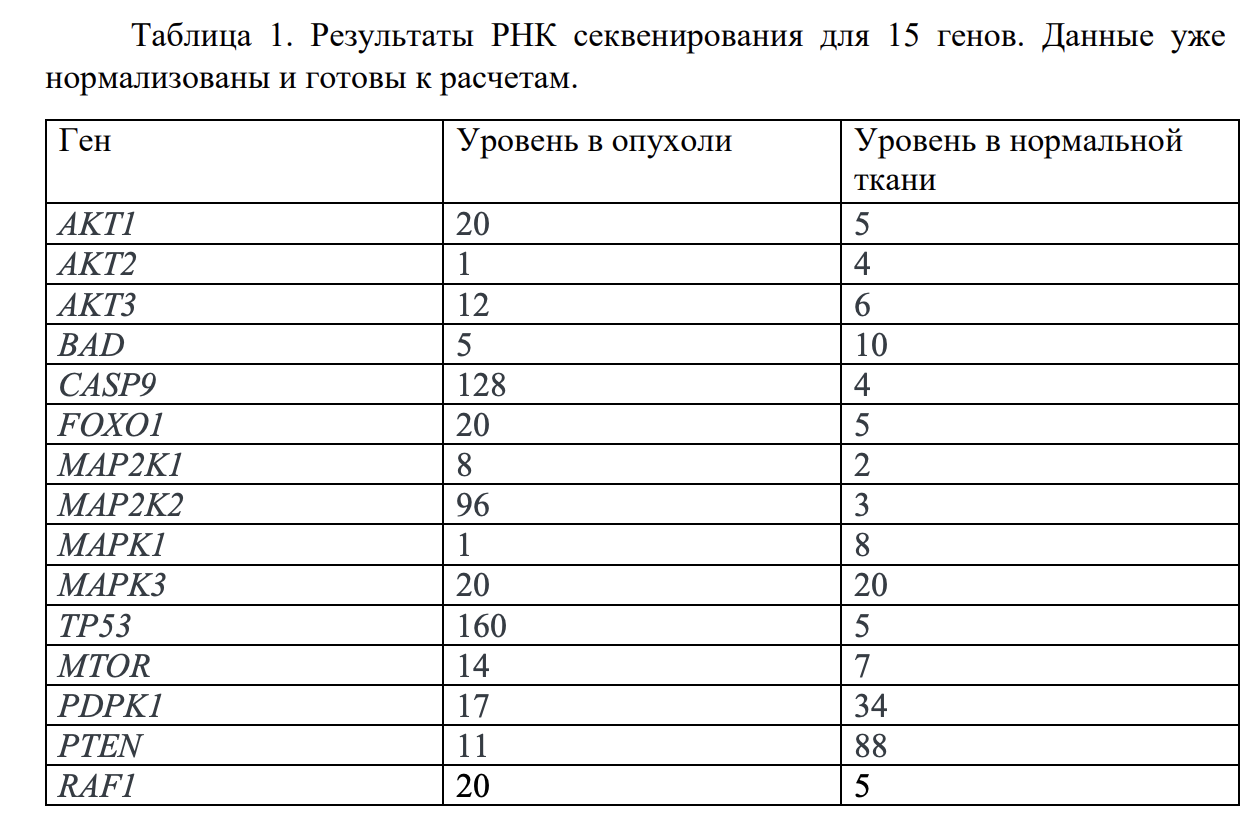
Кроме того, при секвенировании ДНК образца опухоли выявлено, что в генах имеются следующие мутации:
CASP9 c.520G>T p.E174*,
FOXO1 c.725_726insCTGAG p.E242Dfs*2,
MTOR c.5938_5940del p.S1980del.
Для молекулярного пути на рисунке 1:
Задание 1. Какие варианты стартовых вершин возможны?
PDK, PTEN, mTOR
Задание 2. Какую стартовую вершину (вершины) следует выбрать, учитывая биологический эффект пути (подавление выживаемости клетки)? В какое количество вершин из нее можно прийти в случае направленного графа?
PTEN, 5.
Задание 3. Приведите коэффициент для каждой вершины
для PTEN, BAD, FKHR, Caspase 9 коэффициенты равны единице, остальные – минус единице.
Задание 4. Приведите коэффициент и соответствующую вершину для каждого гена из Таблицы 1. Ген может не участвовать в пути.
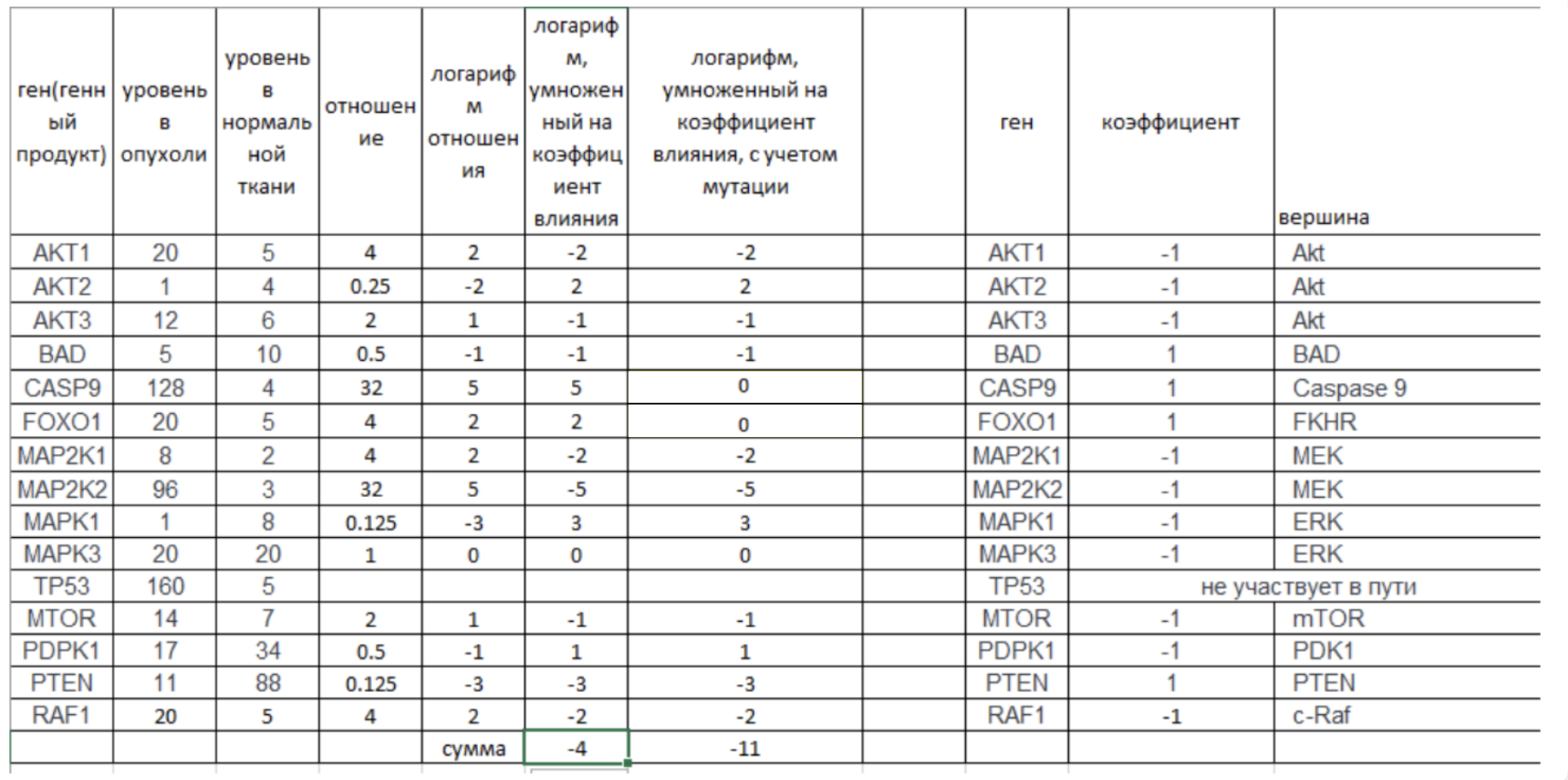
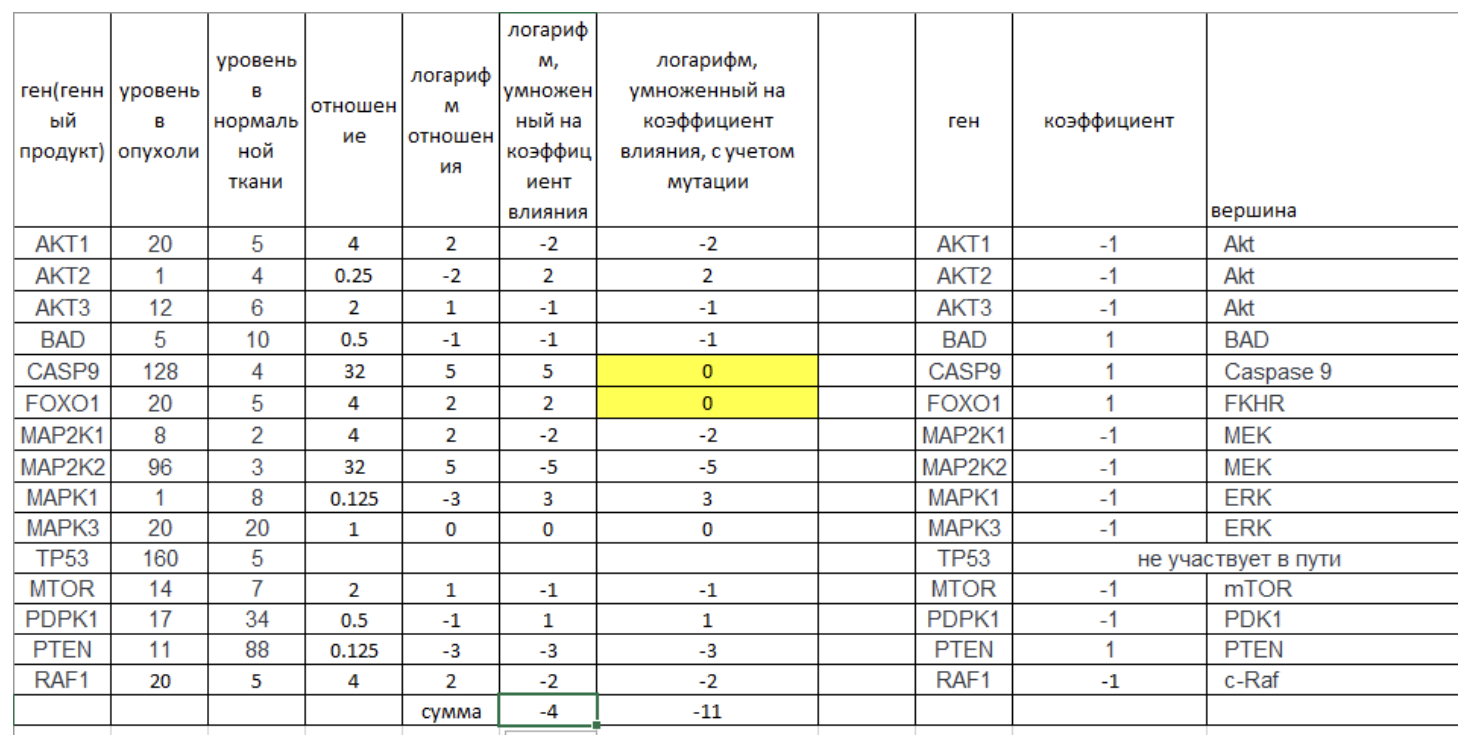
Задание 5. Рассчитайте уровень активности молекулярного пути (число) по экспрессионным данным из Таблицы 1. Также приведите для каждого гена двоичный логарифм отношения опухоль/норма.-4. (Числа простые, расчет доступен без калькулятора).
Задание 6. Предполагая, что мутации, относящиеся к loss-of-function типам, приводят к синтезу нефункционального белка, скорректируйте уровень активации пути. Объясните числовые изменения.
-11.
Объяснение: учитываем только CASP9 c.520G>T p.E174*(nonsense, относится к loss-of-function типам мутаций) и FOXO1 c.725_726insCTGAG p.E242Dfs*2 (инсерция со сдвигом рамки считывания, относится к loss-of-function типам мутаций). Если белок нефункциональный, не имеет значения уровень его экспрессии, поэтому принимаем его нулевым. MTOR c.5938_5940del p.S1980del – делеция, которая не сдвигает рамку считывания, поэтому не относится к loss-of-function типам мутаций.
Задание 7. Рассчитайте процент идентичности для фрагментов белков MAP2K1 и MAP2K2. Процент идентичности равен доле совпавших аминокислот в одинаковых позициях. Предоставьте результаты выравнивания. фрагмент
MAP2K1: MAVGRYPIPPPDAKELELMFGCQVEGDAAETPPRPRTPGRPLSSYGMDSRPPM AIFELLDYIVNEPPPK LPSGVFSLE фрагмент
MAP2K2: LAVGRYPIPPPDAKELEAIFGRPVVDGEEGEPHSISPRPRPPGRPVSGHGMDSRP AMAIFELLDYIVNEPP PKLPNGVFTP
70,7% (58 совпавших аминокислот из 82)

Задание 8. Для расчета уровня активации нескольких путей у нескольких пациентов написан следующий код на языке R:
#create gene - pathway coefficients table
gene_pathway_coefficients <-
data.frame(
pathway1 = c(1, 0, 1, 0, 1, 1),
pathway2 = c(1, -1, -1, 0, 0, 1),
pathway3 = c(1, 0, 1, 0, 0, 0)
)
rownames(gene_pathway_coefficients) <-
paste("Gene", 1:nrow(gene_pathway_coefficients))
print(gene_pathway_coefficients)
#create gene expression table
log2expression_table <- data.frame(matrix(1:12, ncol = 6))
colnames(log2expression_table)<- paste("Gene",1 :ncol(log2expression_table)) rownames(log2expression_table)<-
paste("Patient", 1:nrow(log2expression_table))
print(log2expression_table)
#calculate
pathway activation pathway_activation<-log2expression_table%*%gene_pathway_coefficients
print(pathway_activation)
#sum of activation levels for all pathways and patients
sum(pathway_activation)
Где ошибка, что надо исправить для верной работы кода? Поясните словами и приведите исправленный код. Числовые данные исправлять нельзя. Исправление надо выполнить в пределах 1 существующей строки. Задание выполнять без запуска кода.
Надо исправить класс переменных с датафреймов на матрицы, так как используется матричное умножение.
pathway_activation <- as.matrix(log2expression_table) %*% as.matrix(gene_pathway_coefficients) #исправленная строчка
Задание 9. Какой будет уровень активации пути pathway3 для пациента Patient 1? Какое число напечатает правильный код после последней строчки (сумма уровней активаций всех путей)? Задание выполнять без запуска кода. Активацию путей считать аналогично заданию 5 или так, как указано в коде.
6 ; 78.
Задание 10. Являются ли мутации в генах CASP9 и FOXO1 связанными, то есть возникающими у одних и тех же пациентов? Известно, что CASP9 мутирован у 90 пациентов, ген FOXO1 у 23 пациентов. Всего 345 пациента. При этом у 5 пациентов отмечается наличие мутаций обоих генов одновременно. Дайте ответ на основании \chi2 теста (с пояснением). Приведите ожидаемое количество пациентов с мутациями в обоих генах одновременно.
Ответ: нет, не являются, так как наблюдаемое значение меньше ожидаемого. 6 человек.
Ответ: «нет», пояснение дано. Без пояснения ответ не засчитывается. Ожидаемое значение указано верно (6=90*23/345).
Расчет p-value не нужен (и таблицы для него), так как ответ уже следует из того, что наблюдаемое значение меньше ожидаемого (чтобы считать гены статистически значимо связанными требуется, чтобы наблюдаемое значение было больше ожидаемого и p-value<0.05)